1 对象与方法
1.1 研究对象
1.2 研究方法
1.2.1 超声影像组学特征提取与筛选
1.2.2 外周血miRNA-34a表达水平检测
1.2.3 模型建立与评价
1.3 统计学方法
2 结果
2.1 患者基本特征
表1 训练组和验证组乳腺癌NAC患者的临床特征Table 1 Training and validation group clinical characteristics of breast cancer NAC patients |
| 临床特征 | 训练组(n=75) | 验证组(n=18) | t/χ 2值 | P值 |
|---|---|---|---|---|
| miRNA-34a | 0.64±0.20 | 0.63±0.24 | 0.755 | 0.452 |
| 年龄/岁 | 52.29±4.35 | 49.56±7.33 | 1.520 | 0.145 |
| 肿瘤最大径/mm | 16.75±8.49 | 16.83±7.87 | 0.010 | 0.989 |
| ER/n(%) | 0.640 | 0.425 | ||
| 阴性 | 38(50.67) | 11(61.11) | ||
| 阳性 | 37(49.33) | 7(38.89) | ||
| PR/n(%) | 1.450 | 0.229 | ||
| 阴性 | 41(54.67) | 7(38.89) | ||
| 阳性 | 34(45.33) | 11(61.11) | ||
| HER2/n(%) | 0.010 | 0.931 | ||
| 阴性 | 30(40.00) | 7(38.89) | ||
| 阳性 | 45(60.00) | 11(61.11) | ||
| 钙化/n(%) | 0.320 | 0.571 | ||
| 无 | 55(73.33) | 12(66.67) | ||
| 有 | 20(26.67) | 6(33.33) | ||
| 彩色多普勒血流成像/n(%) | 0.100 | 0.752 | ||
| 无 | 47(62.67) | 12(66.67) | ||
| 有 | 28(37.33) | 6(33.33) | ||
| BI-RADS/n(%) | 0.120 | 0.724 | ||
| 4b及以上 | 69(92.00) | 17(94.44) | ||
| 4a | 6(8.00) | 1(5.56) |
2.2 miRNA-34a表达水平分析
表2 训练组和验证组乳腺癌NAC患者pCR及非pCR的临床特征比较Table 2 Comparison of clinical characteristics between pCR and non-pCR breast cancer NAC patients in the training and validation groups |
| 临床特征 | 训练组 | 验证组 | P单因素分析 | P多因素分析 | |||
|---|---|---|---|---|---|---|---|
| Non-pCR(n=31) | pCR(n=44) | Non-pCR(n=8) | pCR(n=10) | ||||
| miRNA-34a | 0.44±0.10 | 0.77±0.13 | 0.43±0.17 | 0.80±0.15 | 0.001 | 0.015 | |
| 年龄 | 53.16±5.85 | 51.68±2.77 | 49.00±10.88 | 50.00±2.98 | 0.170 | ||
| 肿瘤最大径/mm | 17.84±7.10 | 15.98±9.34 | 22.35±4.52 | 12.41±7.22 | 0.357 | ||
| ER/n(%) | 0.613 | ||||||
| 阴性 | 12(38.71) | 28(63.64) | 3(3/8) | 6(6/10) | |||
| 阳性 | 19(61.29) | 16(36.36) | 5(5/8) | 4(4/10) | |||
| PR/n(%) | 0.019 | 0.719 | |||||
| 阴性 | 19(61.29) | 17(38.64) | 6(6/8) | 6(6/10) | |||
| 阳性 | 12(38.71) | 27(61.36) | 2(2/8) | 4(4/10) | |||
| HER2/n(%) | 0.080 | ||||||
| 阴性 | 14(45.16) | 15(34.09) | 4(4/8) | 4(4/10) | |||
| 阳性 | 17(54.84) | 29(65.91) | 4(4/8) | 6(6/10) | |||
| 钙化/n(%) | 0.080 | ||||||
| 无 | 14(45.16) | 15(34.09) | 4(4/8) | 4(4/10) | |||
| 有 | 17(54.84) | 29(65.91) | 4(4/8) | 6(6/10) | |||
| 彩色多普勒血流成像/n(%) | 0.014 | 0.741 | |||||
| 0 | 19(61.29) | 16(36.36) | 4(4/8) | 7(7/10) | |||
| 1 | 12(38.71) | 28(63.64) | 4(4/8) | 3(3/10) | |||
| BI-RADS/n(%) | 0.280 | ||||||
| 4a | 1(3.23) | 5(11.36) | 1(1/8) | 0 | |||
| 4b及以上 | 30(96.77) | 39(88.64) | 7(7/8) | 10(10/10) | |||
注:单因素和多因素Logistic分析基于训练组。 |
2.3 临床模型构建及诊断表现
表3 不同模型的诊断表现Table 3 Diagnostic performance of different models |
| 组 别 | 模型名称 | AUC(95%CI) | 灵敏度 | 特异度 | 准确度 | 精确度 | F1值 |
|---|---|---|---|---|---|---|---|
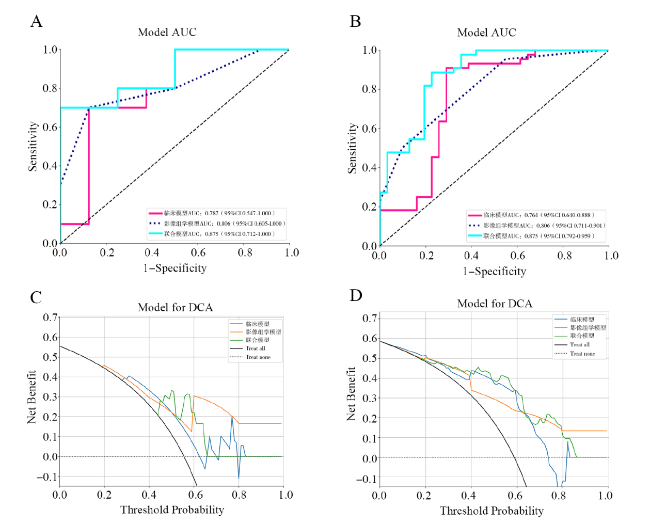
| 训练组 | 临床模型 | 0.787(0.547~1.000) | 0.600 | 0.875 | 0.722 | 0.857 | 0.706 |
| 超声影像组学模型 | 0.806(0.605~1.000) | 0.300 | 1.000 | 0.611 | 1.000 | 0.462 | |
| 联合模型 | 0.875(0.712~1.000) | 0.600 | 1.000 | 0.778 | 1.000 | 0.750 | |
| 验证组 | 临床模型 | 0.764(0.640~0.888) | 0.886 | 0.710 | 0.813 | 0.812 | 0.848 |
| 超声影像组学模型 | 0.806(0.711~0.901) | 0.727 | 0.677 | 0.707 | 0.762 | 0.744 | |
| 联合模型 | 0.875(0.792~0.959) | 0.864 | 0.774 | 0.827 | 0.844 | 0.854 |
2.4 超声影像组学模型构建及诊断表现
2.5 联合模型诊断表现
图1 临床模型、超声影像组学模型、联合模型诊断表现注:A、C为训练组,B、D为验证组。 Figure 1 Diagnostic performance of the clinical model, ultrasound radiomics model and joint model |
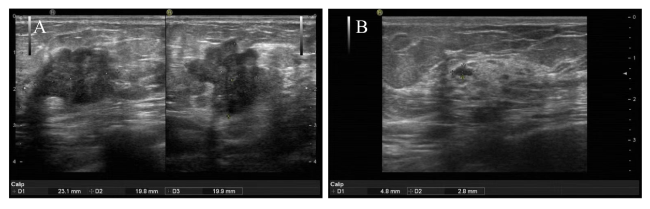
图2 一例联合模型预测乳腺癌NAC患者pCR注:A为NAC前乳腺癌病灶,B为NAC 3个周期后乳腺癌病灶;患者女,55岁,NAC前病灶大小约23.1 mm×19.8 mm×19.9 mm,联合模型预测达到pCR概率为0.857,在接受NAC 3个周期后,病灶大小4.8 mm×2.8 mm,达到pCR。 Figure 2 Joint model for predicting pathological complete response in one case of breast cancer patient after neoadjuvant chemotherapy |