对象与方法
一、研究对象
二、主要试剂
三、实验仪器
四、实验方法
五、统计学处理
结果
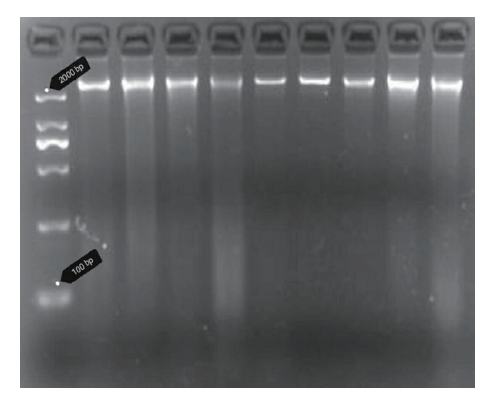
一、提取的粪便DNA琼脂糖凝胶电泳图
二、3组粪便SEPT-9和miRNA-34b/c基因的甲基化阳性率比较
表1 MS-HRM法检测粪便SEPT-9和miRNA-34b/c基因的甲基化阳性率[例(%)] |
| 组 别 | 例数 | SEPT-9 | miRNA-34b/c | 联合检测 | χ2值 | P 值 |
|---|---|---|---|---|---|---|
| 大肠癌组 | 35 | 11(68.6)a | 25(71.4)a | 31(88.6) | χ2SEPT-9 = 5.143 χ2miRNA-34b/c = 4.167 | PSEPT-9 = 0.016 PmiRNA-34b/c = 0.031 |
| 大肠腺瘤组 | 47 | 27(57.4)a | 30(63.8)a | 36(76.6) | χ2SEPT-9 = 7.111 χ2miRNA-34b/c = 4.167 | PSEPT-9 = 0.004 P = 0.031 |
| 正常对照组 | 52 | 5(9.6) | 6(11.5) | 7(13.5) | χ2SEPT-9 = 0.500 χ2miRNA-34b/c = 0 | P = 0.500 P = 1.000 |
注:与正常对照组比较,aP < 0.05/3 |