材料与方法
一、标本来源
二、方法
1. 取样
2. DNA的提取及建库
3. 数据处理及分析
4. 筛选高频基因和驱动基因
5. 肿瘤异质性及亚克隆分析
结果
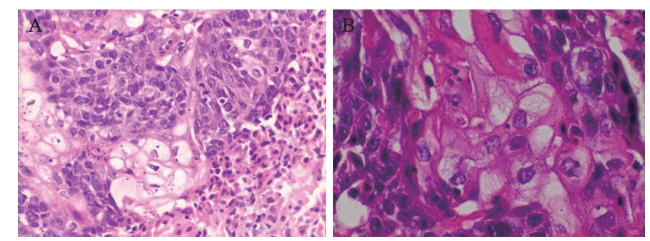
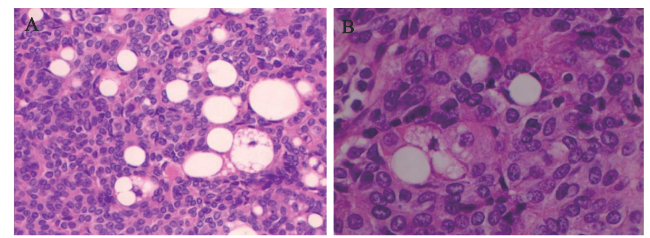
一、病理结果
二、全外显子组测序及生物信息学分析结果
表1 全外显子测序结果数据比对统计情况总览 |
| 样品名 | Clean reads/条 | 比对上参考基因组的reads/条 | 比对 率/% | 重复 reads/条 | 重复rates 比例/% | 探针捕获效率/% | 目标区域的平均测序深度/% | 目标区域的覆盖 度/% | 目标区域深度≥10倍的覆盖区域比例/% |
|---|---|---|---|---|---|---|---|---|---|
| 腮腺SC组织 | 175 543 260 | 165 903 139 | 94.51 | 48 313 747 | 29.12 | 75.57 | 210.68 | 97.17 | 96.29 |
| 腮腺SC癌旁组织 | 141 775 014 | 134 174 396 | 94.64 | 32 465 710 | 24.20 | 74.39 | 179.91 | 97.16 | 96.33 |
| 头皮SA组织 | 136 541 998 | 129 113 256 | 94.56 | 31 182 127 | 24.15 | 77.50 | 177.78 | 97.09 | 96.02 |
| 头皮SA瘤旁组织 | 102 327 688 | 96 319 832 | 94.13 | 22 671 397 | 23.54 | 77.65 | 133.71 | 97.10 | 95.88 |
表2 驱动基因突变筛选结果 |
| 基因名称 | 染色体 | 染色体上的位置 | 参考 碱基 | 突变碱基 | 突变功能 | CGC数据库中对应癌种 | 驱动基因 |
|---|---|---|---|---|---|---|---|
| ABL1 | chr9 | 1.34E+08 | A | G | 错义突变 | 慢性髓细胞性白血病、淋巴细胞白血病、T淋巴细胞-急性淋巴细胞白血病 | 高置信驱动基因 |
| ACTB | chr7 | 5568884 | AGA | — | 框内缺失突变 | — | 候选驱动基因 |
| ACVR2A | chr2 | 1.49E+08 | A | — | 移码缺失突变 | — | 候选驱动基因 |
| AHCTF1 | chr1 | 2.47E+08 | T | G | 错义突变 | — | 候选驱动基因 |
| ASPM | chr1 | 1.97E+08 | T | — | 移码缺失突变 | — | 高置信驱动基因 |
| ATF7IP | chr12 | 14633943 | C | A | 错义突变 | — | 候选驱动基因 |
| BAP1 | chr3 | 52441291 | — | T | 移码插入突变 | 葡萄膜黑色素瘤、乳腺癌、非小细胞肺癌、肾细胞癌 | 高置信驱动基因 |
| CDH1 | chr16 | 68835781 | C | — | 移码缺失突变 | 小叶乳腺癌;胃癌 | 高置信驱动基因 |
| CDKN2A | chr9 | 21974759 | C | — | 移码缺失突变 | 黑色素瘤、多种其他肿瘤类型 | 高置信驱动基因 |
| CHD4 | chr12 | 6707111 | C | T | 错义突变 | 子宫浆液性癌 | 高置信驱动基因 |
| CLIP1 | chr12 | 1.23E+08 | G | A | 错义突变 | 斯皮茨痣样黑色素瘤 | 高置信驱动基因 |
| CNOT1 | chr16 | 58577532 | — | T | 移码插入突变 | — | 候选驱动基因 |
| CNOT3 | chr19 | 54649671 | T | — | 移码缺失突变 | T淋巴细胞-急性淋巴细胞白血病 | 候选驱动基因 |
| COL18A1 | chr21 | 46924438 | CCCCCA | — | 框内缺失突变 | — | 高置信驱动基因 |
| COL18A1 | chr21 | 46876169 | C | T | 错义突变 | — | 高置信驱动基因 |
| CREB1 | chr2 | 2.08E+08 | G | T | 错义突变 | 透明细胞肉瘤、血管瘤样纤维组织细胞瘤 | 高置信驱动基因 |
| CYC1 | chr8 | 1.45E+08 | C | — | 移码缺失突变 | — | 候选驱动基因 |
| DDX3X | chrX | 41193874 | — | G | 移码插入突变 | 慢性淋巴细胞白血病、髓母细胞瘤 | 候选驱动基因 |
| DIDO1 | chr20 | 61510959 | G | A | 错义突变 | — | 候选驱动基因 |
| DMD | chrX | 32235050 | A | G | 错义突变 | — | 高置信驱动基因 |
| DNM3 | chr1 | 1.72E+08 | C | A | 错义突变 | — | 候选驱动基因 |
| DNM3 | chr1 | 1.72E+08 | C | T | 错义突变 | — | 候选驱动基因 |
| DST | chr6 | 56505257 | A | G | 错义突变 | — | 候选驱动基因 |
| EEF1A1 | chr6 | 74229650 | T | C | 错义突变 | — | 高置信驱动基因 |
| EEF1A1 | chr6 | 74228902 | G | A | 错义突变 | — | 高置信驱动基因 |
| FLNA | chrX | 1.54E+08 | T | C | 错义突变 | — | 候选驱动基因 |
| HUWE1 | chrX | 53577645 | C | — | 移码缺失突变 | — | 高置信驱动基因 |
| JAK2 | chr9 | 5066768 | T | — | 移码缺失突变 | 急性淋巴细胞白血病、急性髓系白血病、骨髓增殖性肿瘤、慢性髓细胞性白血病 | 高置信驱动基因 |
| LTBP1 | chr2 | 33525635 | G | A | 错义突变 | — | 候选驱动基因 |
| LZTR1 | chr22 | 21351194 | A | G | 错义突变 | 胶质母细胞瘤 | 高置信驱动基因 |
| MAST2 | chr1 | 46496749 | C | T | 无义突变 | — | 高置信驱动基因 |
| MDN1 | chr6 | 90399738 | A | G | 错义突变 | — | 候选驱动基因 |
| NCOA3 | chr20 | 46279887 | — | CAA | 框内插入突变 | — | 高置信驱动基因 |
| NCOR1 | chr17 | 15995261 | G | A | 无义突变 | 乳腺癌 | 高置信驱动基因 |
| NR4A2 | chr2 | 1.57E+08 | T | — | 移码缺失突变 | — | 高置信驱动基因 |
| NUP98 | chr11 | 3726447 | G | A | 错义突变 | 急性髓系白血病 | 高置信驱动基因 |
| P4HB | chr17 | 79817126 | C | T | 错义突变 | — | 候选驱动基因 |
| PLEC | chr8 | 1.45E+08 | C | G | 错义突变 | — | 高置信驱动基因 |
| PLEC | chr8 | 1.45E+08 | C | T | 错义突变 | — | 高置信驱动基因 |
| PLEC | chr8 | 1.45E+08 | G | A | 错义突变 | — | 高置信驱动基因 |
| PPFIA4 | chr1 | 2.03E+08 | C | T | 错义突变 | — | 候选驱动基因 |
| RAG1 | chr11 | 36596125 | AAG | — | 框内缺失突变 | — | 候选驱动基因 |
| RNF43 | chr17 | 56435161 | C | — | 移码缺失突变 | 胆管癌、卵巢癌、胰腺癌 | 高置信驱动基因 |
| RNF43 | chr17 | 56435193 | AG | — | 移码缺失突变 | 胆管癌、卵巢癌、胰腺癌 | 高置信驱动基因 |
| RPL22 | chr1 | 6257785 | T | — | 移码缺失突变 | 急性髓系白血病、慢性髓细胞性白血病 | 高置信驱动基因 |
| RXRA | chr9 | 1.37E+08 | G | A | 错义突变 | — | 高置信驱动基因 |
| SETD2 | chr3 | 47164922 | G | A | 错义突变 | 肾透明细胞癌 | 高置信驱动基因 |
| SPTAN1 | chr9 | 1.31E+08 | T | C | 错义突变 | — | 候选驱动基因 |
| SRRM2 | chr16 | 2816326 | C | T | 错义突变 | — | 候选驱动基因 |
| STAT3 | chr17 | 40491387 | G | A | 错义突变 | T细胞大颗粒淋巴细胞白血病 | 高置信驱动基因 |
| TBL1XR1 | chr3 | 1.77E+08 | T | — | 移码缺失突变 | 脾边缘区淋巴瘤、原发性中枢神经系统淋巴瘤、结直肠癌、胆囊癌 | 高置信驱动基因 |
| TCF7L2 | chr10 | 1.15E+08 | A | — | 移码缺失突变 | 结直肠癌 | 高置信驱动基因 |
| TET2 | chr4 | 1.06E+08 | A | T | 错义突变 | 骨髓增生异常综合征 | 高置信驱动基因 |
| TTK | chr6 | 80751897 | A | — | 移码缺失突变 | — | 候选驱动基因 |
| UHRF1BP1L | chr12 | 1E+08 | G | T | 错义突变 | — | 高置信驱动基因 |
| ZFHX3 | chr16 | 72993474 | A | G | 错义突变 | 子宫内膜癌、胃癌、前列腺癌 | 高置信驱动基因 |
| ZMYND8 | chr20 | 45849979 | T | A | 无义突变 | — | 候选驱动基因 |