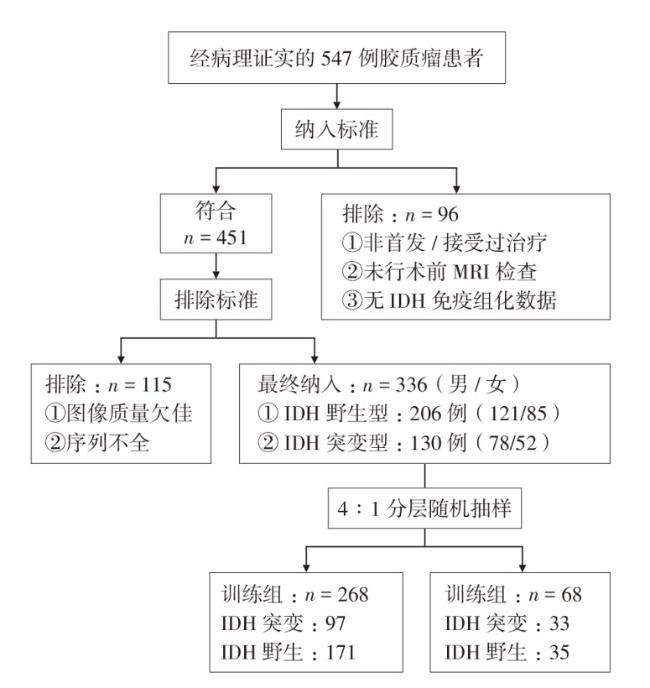
1 对象与方法
1.1 研究对象
1.2 检查方法
1.3 图像分析
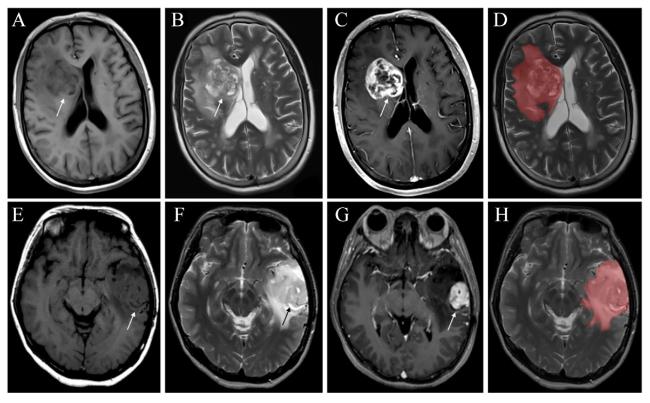
图1 一例胶质母细胞瘤及弥漫性星形细胞瘤患者的MRI图像及ROI勾画注:A~D为胶质母细胞瘤,IDH野生型,患者男,68岁。A~C分别为T1WI、T2WI及T1WI增强,病灶位于右侧侧脑室旁,呈长T1WI长T2WI混杂信号,边界欠清,周围脑实质水肿明显,增强扫描可见明显环形强化。D为以轴向T2WI图像为基础,在每个层面上勾画ROI,覆盖整个肿瘤(包括坏死、囊变区)及其瘤周水肿区域。E~H为弥漫性星形细胞瘤,IDH突变型,患者男,72岁。E~G分别为T1WI、T2WI及T1WI增强,病灶位于左侧颞叶,与胶质母细胞瘤表现相似,肉眼难以区分。H为ROI勾画方法,与D一致。 Figure 1 MRI images and ROI delineation of a patient with glioblastoma and diffuse astrocytoma |
1.4 特征提取及筛选
1.5 模型建立及评估
1.6 统计学方法
2 结果
2.1 不同分组间患者年龄和性别的比较
表1 IDH突变组与IDH野生组患者基本资料Table 1 Basic information of patients in the IDH mutant group and the IDH wild-type group |
| 项 目 | IDH野生组(n=206) | IDH突变组(n=130) | t/ χ 2值 | P值 |
|---|---|---|---|---|
| 年龄/岁 | 49.17±14.28 | 42.25±10.85 | -5.029 | <0.001 |
| 男性/n(%) | 121(58.7) | 78(60.0) | 0.530 | 0.819 |
| WHO分级/n(%) | 72.750 | <0.001 | ||
| 低级别 | 36(17.5) | 82(63.1) | ||
| 高级别 | 170(82.5) | 48(36.9) | ||
表2 训练组与测试组患者基本资料Table 2 Basic information of patients in the training group and the test group |
| 项 目 | 训练组(n=268) | 测试组(n=68) | t/ χ 2值 | P值 |
|---|---|---|---|---|
| 年龄/岁 | 45.92±12.60 | 46.28±13.05 | -0.189 | 0.850 |
| 男性/n(%) | 158(59.0) | 41(60.3) | 0.032 | 0.860 |
| IDH/n(%) | 0.015 | 0.902 | ||
| 野生型 | 171(63.8) | 35(51.5) | ||
| 突变型 | 97(36.2) | 33(48.5) | ||
| WHO分级/n(%) | 2.789 | 0.095 | ||
| 低级别 | 87(32.5) | 31(45.6) | ||
| 高级别 | 181(67.5) | 37(54.4) |
2.2 特征参数
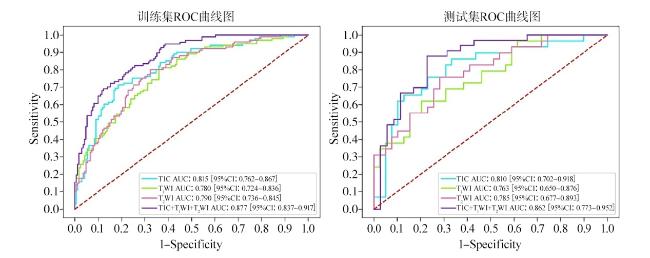
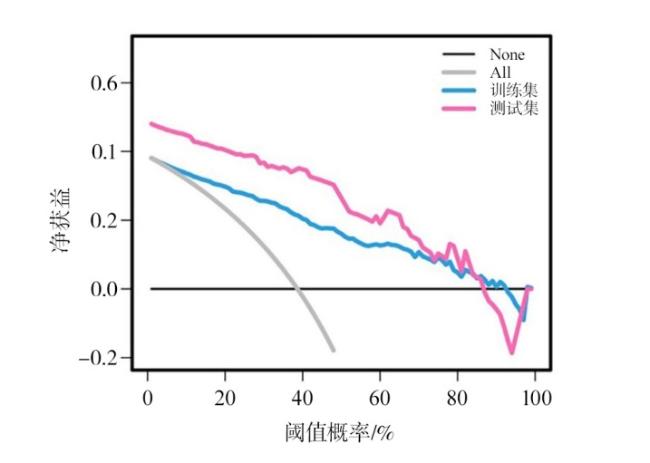
2.3 常规MRI影像组学模型预测胶质瘤IDH突变状态的效能
表3 基于常规MRI影像组学模型预测IDH突变状态的效能Table 3 Performance of conventional MRI-based radiomics models in predicting IDH mutation status |
| 序 列 | AUC(95%CI) | 准确率 | |||
|---|---|---|---|---|---|
| 训练组(n=268) | 测试组(n=68) | 训练组(n=268) | 测试组(n=68) | ||
| T1WI | 0.780(0.724,0.836) | 0.763(0.650,0.876) | 0.798 | 0.794 | |
| T2WI | 0.790(0.736,0.845) | 0.785(0.677,0.893) | 0.836 | 0.809 | |
| 增强 | 0.815(0.762,0.867) | 0.810(0.702,0.918) | 0.847 | 0.809 | |
| 联合 | 0.877(0.837,0.917) | 0.862(0.773,0.952) | 0.877 | 0.853 | |
注:增强代表T1WI增强序列;联合代表T1WI、T2WI及增强序列。 |
表4 基于常规MRI影像组学模型预测IDH突变状态的应用价值Table 4 Application value of conventional MRI-based radiomics model in predicting IDH mutation status |
| 序 列 | 灵敏度 | 特异度 | 阳性预测值 | 阴性预测值 | |||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| 训练组 | 测试组 | 训练组 | 测试组 | 训练组 | 测试组 | 训练组 | 测试组 | ||||
| T1WI | 0.804 | 0.794 | 0.795 | 0.771 | 0.684 | 0.771 | 0.877 | 0.818 | |||
| T2WI | 0.814 | 0.818 | 0.847 | 0.800 | 0.755 | 0.794 | 0.896 | 0.824 | |||
| 增强 | 0.804 | 0.864 | 0.825 | 0.788 | 0.769 | 0.813 | 0.896 | 0.805 | |||
| 联合 | 0.877 | 0.862 | 0.888 | 0.857 | 0.814 | 0.848 | 0.916 | 0.857 | |||
注:增强代表T1WI增强序列;联合代表T1WI、T2WI及增强序列。 |
表5 不同模型之间AUC两两比较Table 5 Pairwise comparison of AUC among different models |
| 模型对比 | AUC | Z值 | 原始P值 |
|---|---|---|---|
| T1WI vs. T2WI | 0.780 vs.0.790 | 0.32 | 0.749 |
| T1WI vs. 增强 | 0.780 vs.0.815 | 1.15 | 0.249 |
| T2WI vs. 增强 | 0.790 vs.0.815 | 0.82 | 0.413 |
| 联合vs. T1WI | 0.877 vs.0.780 | 3.12 | 0.002 |
| 联合vs. T2WI | 0.877 vs.0.790 | 2.89 | 0.004 |
| 联合vs. 增强 | 0.877 vs.0.815 | 2.35 | 0.018 |
3 讨论